✓ 24/7 automatic processing of online orders
✓ Knowledgeable and professional Product & Technical Support
✓ Fast and reliable (re)-ordering
RNeasy Fibrous Tissue Mini Kit (50)
Cat no. / ID. 74704
✓ 24/7 automatic processing of online orders
✓ Knowledgeable and professional Product & Technical Support
✓ Fast and reliable (re)-ordering
Features
- Optimized protocols for fibrous tissues
- High yields of total RNA
- High-quality RNA for all downstream applications
- Removal of genomic DNA using DNase I
Product Details
The RNeasy Fibrous Tissue Mini Kit includes proteinase K for removing abundant protein in fiber-rich tissue samples, and RNeasy spin columns for purifying up to 100 µg of high-quality RNA. The kit can be automated on the QIAcube Connect. Fibrous tissue samples can be conveniently stabilized using RNAprotect Tissue Reagent or Allprotect Tissue Reagent, and efficiently disrupted using a TissueRuptor or TissueLyser system. For larger samples, the RNeasy Fibrous Tissue Midi Kit (spin-column binding capacity of 1 mg RNA) is also available.
Performance
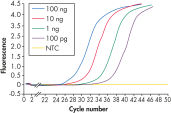
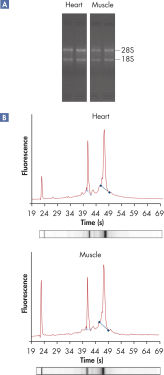
RNeasy Fibrous Tissue Kits are optimized for use with fiber-rich tissues and the RNeasy Fibrous Tissue procedure ensures easy and efficient isolation of high-quality total RNA (see figure " High-quality RNA from fiber-rich tissues") suitable for any downstream application, including array analysis and real-time RT-PCR (see figures " Real-time gene expression analysis in a variety of fibrous tissues" and " LightCycler analysis of high-quality RNA from heart"). The RNeasy Fibrous Tissue Mini Kit ensures higher yields of RNA from fiber-rich tissue compared with other silica-based methods (see table).
| Rat tissue (10 mg) |
RNA yield* (µg) with RNeasy Fibrous Tissue Mini Kit |
RNA yield* (µg) with silica kit (Supplier AV |
RNA yield* (µg) with silica kit (Supplier R) |
RNA yield* (µg) with silica kit (Supplier S) |
|---|---|---|---|---|
| Heart | 8.2 | 3.5 | <0.5 | 2.5 |
| Muscle | 5.3 | 0.5 | 2.0† | 2.2 |
| Skin | 6.0 | Not determined | Not determined | Not determined |
| Esophagus | 13.9 | Not determined | Not determined | Not determined |
See figures
Principle
RNeasy technology simplifies total RNA isolation by combining the stringency of guanidine-isothiocyanate lysis with the speed and purity of silica-membrane purification. RNeasy Fibrous Tissue Kits are optimized for use with fiber-rich tissues, such as skeletal muscle, heart, and aorta, tissues difficult to lyse due to the abundance of contractile proteins, connective tissue, and collagen. The RNeasy Fibrous Tissue protocol integrates proteinase K and RNase-free DNase digestion steps during RNA isolation to break down proteins and genomic DNA for higher yields of high-quality total RNA. Since the RNeasy procedures enrich for mRNA and other RNA species >200 nucleotides, the total RNA yield does not include 5S rRNA, tRNA, and other low-molecular-weight RNAs, which make up 15-20% of total cellular RNA.
Procedure
Samples of fibrous tissue from 0.5–30 mg are lysed in a guanidine-isothiocyanate buffer. After dilution of the lysate, the sample is treated with proteinase K. Debris is pelleted by centrifugation. Ethanol is then added to the cleared lysate, and RNA is bound to the RNeasy silica membrane. Traces of DNA that may copurify are removed by a DNase treatment on the RNeasy spin column. DNase and any contaminants are washed away, and up to 100 µg high-quality total RNA is eluted in 30–100 µl RNase-free water (see figure " RNeasy Fibrous Tissue Mini procedure").
See figures
Applications
RNeasy Fibrous Tissue Kits provide high-quality total RNA from fiber-rich samples that is suitable for any downstream application, including array analysis and real-time RT-PCR.
Supporting data and figures
High-quality RNA from fiber-rich tissues.
Specifications
| Features | Specifications |
|---|---|
| applications | PCR, real-time PCR, microarray |
| elutionvolume | 30–100 µl |
| format | Spin column |
| timeperrunorperprep | 55 minutes |
| purificationoftotalrnamirnapolyamrnadnaorprotein | RNA |
| processing | Manual |
| sampleamount | 0.5–30 mg |
| technology | Silica technology |
| mainsampletype | Fibre-rich tissue samples |
| yield | 5–12 µg |